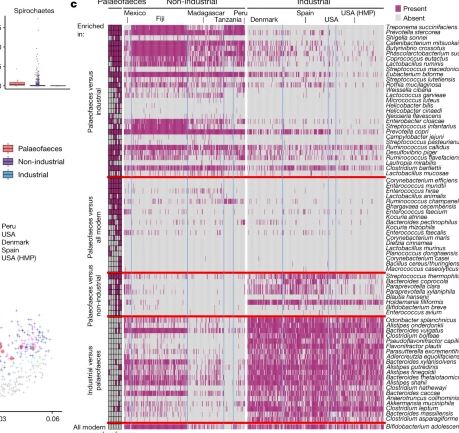
A perda da diversidade microbiana intestinal em populações industriais está associada a doenças crônicas 7, ressaltando a importância de estudar nosso microbioma intestinal ancestral. No entanto, relativamente pouco se sabe sobre a composição dos microbiomas intestinais pré-industriais. Aqui realizamos uma montagem de novo em grande escala de genomas microbianos de palaeofaeces. A partir de oito amostras de palaeofaeces humanas autenticadas (1.000–2.000 anos de idade) com DNA bem preservado do sudoeste dos EUA e do México, reconstruímos 498 genomas microbianos de média e alta qualidade. Entre os 181 genomas com as evidências mais fortes de serem antigos e de origem no intestino humano, 39% representam caixas de genoma em nível de espécie anteriormente não descritas. A datação de dicas sugere um cronograma de diversificação aproximado para o simbionte humano-chave Methanobrevibacter smithii. Em comparação com 789 amostras atuais de microbiomas intestinais humanos de oito países, as amostras de palaeofaeces são mais semelhantes a microbiomas intestinais humanos não industrializados do que industrializados. O perfil funcional das amostras de palaeofaeces revela uma abundância significativamente menor de genes de resistência a antibióticos e de degradação de mucina, bem como enriquecimento de elementos genéticos móveis em relação aos microbiomas intestinais industriais. Este estudo facilita a descoberta e caracterização de microrganismos intestinais anteriormente não descritos de microbiomas antigos e a investigação da história evolutiva da microbiota intestinal humana por meio da reconstrução do genoma a partir de paleofemas.
Loss of gut microbial diversity in industrial populations is associated with chronic diseases7, underscoring the importance of studying our ancestral gut microbiome. However, relatively little is known about the composition of pre-industrial gut microbiomes. Here we performed a large-scale de novo assembly of microbial genomes from palaeofaeces. From eight authenticated human palaeofaeces samples (1,000–2,000 years old) with well-preserved DNA from southwestern USA and Mexico, we reconstructed 498 medium- and high-quality microbial genomes. Among the 181 genomes with the strongest evidence of being ancient and of human gut origin, 39% represent previously undescribed species-level genome bins. Tip dating suggests an approximate diversification timeline for the key human symbiont Methanobrevibacter smithii. In comparison to 789 present-day human gut microbiome samples from eight countries, the palaeofaeces samples are more similar to non-industrialized than industrialized human gut microbiomes. Functional profiling of the palaeofaeces samples reveals a markedly lower abundance of antibiotic-resistance and mucin-degrading genes, as well as enrichment of mobile genetic elements relative to industrial gut microbiomes. This study facilitates the discovery and characterization of previously undescribed gut microorganisms from ancient microbiomes and the investigation of the evolutionary history of the human gut microbiota through genome reconstruction from palaeofaeces.